-
-
Notifications
You must be signed in to change notification settings - Fork 266
RStan Getting Started (Japanese)
この文書はRStan Getting Startedを日本語に翻訳したものです。一部構成を改変しています。
RStanはStanをR から利用するためのインターフェースです。この文書では以下の説明をします:
Stanやその文法のさらなる情報が欲しい場合は以下の公式Webサイトを見てください:
下の説明のほとんどは最新バージョンのRStanに関するものです。Rのバージョンは3.4.0かそれ以降が必要です。必要あれば、ここから最新版のRをインストールできます。
あと、ユーザにはRStudioのバージョン1.2.x以降の使用を強くオススメします。なぜならStanのコーディングを強力に支援する機能があるからです。
RtoolsはRとうまく連携できるC++コンパイラを提供します。Rtoolsは主にGNU makeとGNU gccとUNIXライクなプラットフォームで一般的に使われるコマンド群を含みます。
以降ではWindowsの場合に、C++コンパイラをダウンロードしてインストールする方法を説明します。
- Macの場合、RStan Mac OS X Prerequisite Installation Instructions を見てください。
- Linuxの場合、使っているディストリビューションのパッケージマネージャーを使ってビルドに必要なものと最近のg++かclang++をインストールしてください。
Rtoolsを http://cran.r-project.org/bin/windows/Rtools/ からインストールできます。もし、インストールしたRのバージョンが3.3.xならば、Rtools33.exeをダウンロードして実行してください。もし、バージョンが3.2.xかそれより下ならば、Rtools33.exeを使わないで、インストールしたRのバージョンにあったなかで最も新しい「frozen」になっているRtoolsを選んでください。もし、複数のバージョンのRを使いたいならば、複数のバージョンのRtoolsを入れて使うことも可能です。しかし、Stanのプログラムをコンパイルするために何らかの環境変数をいじる必要があるでしょう。
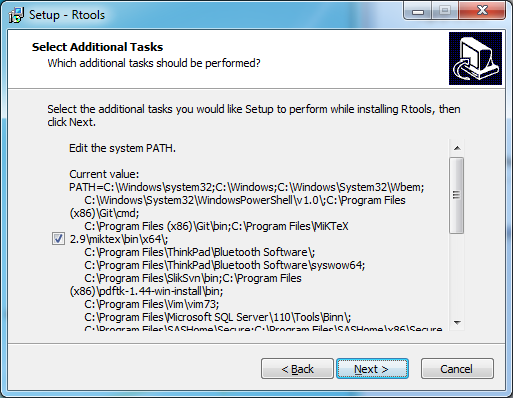
Rtoolsをインストールする際に、環境変数PATHを編集するステップに注意してください。以下の画像で示したように、このオプションにチェックを入れましょう(何もタイプする必要はありません。ただチェックするだけです)。こうすることで、RからC++コンパイラを使えるようになります。
- 新しくRを起動します。
-
Sys.getenv("PATH")を実行して、環境変数PATHにRtoolsが含まれているかチェックします。もし、Rtoolsをc:\\Rtoolsにインストールした場合、以下のような出力が見えるはずです。
> Sys.getenv('PATH')
[1] "c:\\\\Rtools\\\\bin;c:\\\\Rtools\\\\gcc-4.6.3\\\\bin;...
上の出力の例では、はじめの方がc:\\Rtools\\bin and c:\\Rtools\\gcc-4.x-y\\binとなっています。もし、Rtoolsをc:\\Rtoolsにインストールして、これら2つが出てこなかったら、上のRtoolsのインストールの節に書いてあることにきちんと従ったか確かめてください。もし、うまくいかなかったら、選択肢は二つあります。一つ目はRtoolsを再インストールすることで、もう一つは環境変数PATHを手動で編集することです。簡単なので、前者の方をまずはオススメします。
- Rから本当に
g++を呼び出せるかチェックします。例えば、以下のようにしてRからgccのバージョンを見ることができます。
> system('g++ -v')
Using built-in specs.
COLLECT_GCC=c:\Rtools\GCC-46~1.3\bin\G__~1.EXE
COLLECT_LTO_WRAPPER=c:/rtools/gcc-46~1.3/bin/../libexec/gcc/i686-w64-mingw32/4.6.3/lto-wrapper.exe
Target: i686-w64-mingw32
Configured with: /data/gannet/ripley/Sources/mingw-test3/src/gcc/configure --host=i686-w64-mingw32 --build=x86_64-linux-gnu --target=i686-w64-mingw32 --with-sysroot=/data/gannet/ripley/Sources/mingw-test3/mingw32mingw32/mingw32 --prefix=/data/gannet/ripley/Sources/mingw-test3/mingw32mingw32/mingw32 --with-gmp=/data/gannet/ripley/Sources/mingw-test3/mingw32mingw32/prereq_install --with-mpfr=/data/gannet/ripley/Sources/mingw-test3/mingw32mingw32/prereq_install --with-mpc=/data/gannet/ripley/Sources/mingw-test3/mingw32mingw32/prereq_install --disable-shared --enable-static --enable-targets=all --enable-languages=c,c++,fortran --enable-libgomp --enable-sjlj-exceptions --enable-fully-dynamic-string --disable-nls --disable-werror --enable-checking=release --disable-win32-registry --disable-rpath --disable-werror CFLAGS='-O2 -mtune=core2 -fomit-frame-pointer' LDFLAGS=
Thread model: win32
gcc version 4.6.3 20111208 (prerelease) (GCC)
> system('where make')
c:\Rtools\bin\make.exe
以下は必須ではありません。やらなくてもRStanは動くからです。しかし、コンパイル設定のカスタマイズを行うと速度向上が期待できます。
やっとRStanをインストールする準備ができました!以下のステップに順に従うとRStanをインストールできます。
- Rを起動します(R GUIでもターミナル上で起動したRでもオススメしたRStudioを起動して実行してもOKです)。
- rstanパッケージの最新版とそれが依存しているパッケージを以下のように明示的に指定してCRANからインストールします。
# もしhttpsを使ったダウンロードに対応していないならば、httpsの「s」を取って実行してください
install.packages('rstan', repos='https://cloud.r-project.org/', dependencies=TRUE)dependencies=TRUEを省略しないでください。
- Stanがちょうどバージョンアップをしている最中(StanのリリースはされたけどCRANのrstanのバイナリの最新版ができあがっていない状態)の場合には、上記の
install.packages関数を実行する代わりにここを読んでインストールする必要があります。 - それでもうまくいかない場合、ソースからインストールすることを試してもよいかもしれません。
# 注:以下の「4」をビルドに使いたいコア数に置き換えてください
Sys.setenv(MAKEFLAGS = "-j4")
install.packages("rstan", type = "source")-
Rの再起動 インストールの後はたぶんRの再起動が必要です。 そしてrstanパッケージを呼び出す前に以前のバージョンのRStanによって作られた何らかのオブジェクトが勝手にloadされないか確かめてください。
-
Rで以下を実行して、C++が動くか確かめてください。
fx <- inline::cxxfunction( signature(x = "integer", y = "numeric" ) , '
return ScalarReal( INTEGER(x)[0] * REAL(y)[0] ) ;
')
fx( 2L, 5 ) # 10になるはずですパッケージ名は「rstan」(すべて小文字)です、だから library(rstan) でrstanをloadする必要があります。
library(rstan) # スタートアップのメッセージが表示されるスタートアップのメッセージにあるように、もしマルチコアでメモリも十分にあるローカルマシンでrstanを使って並列で推定計算を実行させたい場合には、以下を実行します。
rstan_options(auto_write = TRUE)
options(mc.cores = parallel::detectCores())これらのoptionはそれぞれ、再コンパイルをしなくてすむようにコンパイル後のStanプログラムをハードディスクに保存すること、複数のマルコフ連鎖を並列で実行することを指定しています。
これはGelman et al (2003)の5.5節にある例です。8つの学校における教育の効果を研究したものです。簡単のため、この例を「eight schools」と呼びます。
はじめに、モデルをStanプログラムで書いて8schools.stanという名前で保存します。中身は以下の通りです(ファイルはここで見ることができます)。
data {
int<lower=0> J; // 学校の数
real y[J]; // 推定されている教育の効果
real<lower=0> sigma[J]; // 教育の効果の標準誤差
}
parameters {
real mu;
real<lower=0> tau;
real eta[J];
}
transformed parameters {
real theta[J];
for (j in 1:J)
theta[j] = mu + tau * eta[j];
}
model {
target += normal_lpdf(eta | 0, 1);
target += normal_lpdf(y | theta, sigma);
}このモデルでは、thetaをparametersとして直接的に宣言する代わりに、muとetaを使ってthetaを作りtransformed parametersにしています。このようにパラメータ化することでより効率的にサンプリングできます(詳しい説明はこちらを参照してください)。8schools.stanを作業ディレクトリに置いたとすると、以下のRコードでデータを用意することができ、
schools_dat <- list(J = 8,
y = c(28, 8, -3, 7, -1, 1, 18, 12),
sigma = c(15, 10, 16, 11, 9, 11, 10, 18))以下のRコードでモデルをフィッティングさせることができます。
fit <- stan(file = '8schools.stan', data = schools_dat,
iter = 1000, chains = 4)stan関数のmodel_code引数を使うことでStanのモデルをRコード内の文字列で指定することもできます。しかしながら、これはオススメできません。なぜなら、ファイルを使えばprint文が使えたり、エラーメッセージの中の行番号を参考にすることができるからです。
stan関数から返されたfitオブジェクトはstanfitクラスのS4オブジェクトです。 print・plot・pairsのような関数はフィットした結果と関連しており、このあとのコードを使ってfitの中の結果を取り出すことができます。printはモデルのパラメータに加えてlp__という名前の対数事後確率(log-posterior)の要約を表示します(このあとの出力例を見てください)。他の関数やstanfitクラスの詳細が知りたい場合は、stanfitクラスのヘルプを見てください。
特に、MCMCサンプルを得るにはstanfitオブジェクトに対してextract関数を使います。 extractはstanfitオブジェクトから、興味あるパラメータのarraysのlistもしくは1つのarrayとしてMCMCサンプルを取り出します。さらに、as.array・as.matrix・as.data.frameというS3関数が定義されています(R上でhelp("as.array.stanfit")を使ってヘルプを見てください)。
print(fit)
plot(fit)
pairs(fit, pars = c("mu", "tau", "lp__"))
la <- extract(fit, permuted = TRUE) # arraysのlistを返す
mu <- la$mu
### iterations, chains, parametersの3次元arrayを返す
a <- extract(fit, permuted = FALSE)
### stanfitオブジェクトにS3関数を使う
a2 <- as.array(fit)
m <- as.matrix(fit)
d <- as.data.frame(fit)> print(fit, digits = 1)
Inference for Stan model: 8schools.
4 chains, each with iter=1000; warmup=500; thin=1;
post-warmup draws per chain=500, total post-warmup draws=2000.
mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
mu 8.2 0.2 5.4 -1.9 4.8 8.1 11.3 19.3 480 1
tau 6.8 0.3 6.2 0.3 2.5 5.2 9.2 21.7 425 1
eta[1] 0.4 0.0 1.0 -1.5 -0.3 0.4 1.0 2.2 2000 1
eta[2] 0.0 0.0 0.8 -1.7 -0.6 0.0 0.5 1.7 2000 1
eta[3] -0.2 0.0 1.0 -2.1 -0.9 -0.2 0.4 1.7 2000 1
eta[4] -0.1 0.0 0.9 -1.8 -0.7 -0.1 0.5 1.7 2000 1
eta[5] -0.4 0.0 0.9 -2.1 -1.0 -0.4 0.2 1.4 2000 1
eta[6] -0.2 0.0 0.9 -1.9 -0.8 -0.2 0.4 1.5 1731 1
eta[7] 0.3 0.0 0.9 -1.4 -0.2 0.4 0.9 2.0 1507 1
eta[8] 0.0 0.0 0.9 -1.9 -0.6 0.0 0.7 1.8 1988 1
theta[1] 11.5 0.3 8.8 -2.4 5.9 10.1 15.6 32.9 977 1
theta[2] 7.8 0.1 6.2 -4.7 4.1 7.9 11.6 20.3 2000 1
theta[3] 6.1 0.2 7.7 -11.2 2.1 6.4 10.5 20.2 2000 1
theta[4] 7.6 0.1 6.5 -4.9 3.8 7.8 11.4 21.3 2000 1
theta[5] 5.0 0.1 6.6 -9.3 1.2 5.6 9.3 16.7 2000 1
theta[6] 6.2 0.2 6.7 -8.2 2.2 6.5 10.5 18.5 2000 1
theta[7] 10.8 0.2 7.0 -1.3 6.1 10.1 15.1 26.8 2000 1
theta[8] 8.7 0.2 8.2 -7.3 3.9 8.4 12.8 27.2 1446 1
lp__ -39.5 0.1 2.6 -45.1 -41.2 -39.4 -37.7 -35.1 590 1
Samples were drawn using NUTS(diag_e) at Fri May 5 10:41:43 2017.
For each parameter, n_eff is a crude measure of effective sample size,
and Rhat is the potential scale reduction factor on split chains (at
convergence, Rhat=1).
さらに、BUGS(やJAGS)と同様に、CmdStan(Stanのコマンドラインインターフェース)を使うにはすべてのデータをRのdumpファイルの形式で持っておく必要があります。dumpファイルがある場合、rstanにはread_rdumpという関数があり、これを使って全てのデータを1つのRの名前付きリストとして読み込むことができます。例えば、作業ディレクトリに以下の中身を持つ "8schools.rdump" という名前のファイルがあるとします。
J <- 8
y <- c(28, 8, -3, 7, -1, 1, 18, 12)
sigma <- c(15, 10, 16, 11, 9, 11, 10, 18)そうすると以下のように "8schools.rdump" からデータを読み込むことができます。
schools_dat <- read_rdump('8schools.rdump')dumpファイルはRのsource関数を使って読み込むこともでき、dumpファイル内の各変数はグローバル環境内のオブジェクトとなります。この場合、Stanを実行する時のstan関数のdata引数を省略することができます。stan関数は 8schools.stan のdataブロックにある変数名と同じ名前のオブジェクトを呼び出された環境から探すからです。すなわち、
source('8schools.rdump')
fit <- stan(file = '8schools.stan', iter = 1000, chains = 4)Ratsの例も人気のある例です。 例えば、ここにOpenBUGSバージョンがあり、オリジナルはGelfand et al (1990)です。30匹のラットの体重を1週間ごと5週間にわたって記録したデータです。データは以下の表のようになっており、体重を記録した日付を表すのにxを使っています。この例を試すのにリンク先のデータとモデルのコードを使うことができます(rats.txtとrats.stan)。
| Rat | x=8 | x=15 | x=22 | x=29 | x=36 | Rat | x=8 | x=15 | x=22 | x=29 | x=36 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 151 | 199 | 246 | 283 | 320 | 16 | 160 | 207 | 248 | 288 | 324 | |
| 2 | 145 | 199 | 249 | 293 | 354 | 17 | 142 | 187 | 234 | 280 | 316 | |
| 3 | 147 | 214 | 263 | 312 | 328 | 18 | 156 | 203 | 243 | 283 | 317 | |
| 4 | 155 | 200 | 237 | 272 | 297 | 19 | 157 | 212 | 259 | 307 | 336 | |
| 5 | 135 | 188 | 230 | 280 | 323 | 20 | 152 | 203 | 246 | 286 | 321 | |
| 6 | 159 | 210 | 252 | 298 | 331 | 21 | 154 | 205 | 253 | 298 | 334 | |
| 7 | 141 | 189 | 231 | 275 | 305 | 22 | 139 | 190 | 225 | 267 | 302 | |
| 8 | 159 | 201 | 248 | 297 | 338 | 23 | 146 | 191 | 229 | 272 | 302 | |
| 9 | 177 | 236 | 285 | 350 | 376 | 24 | 157 | 211 | 250 | 285 | 323 | |
| 10 | 134 | 182 | 220 | 260 | 296 | 25 | 132 | 185 | 237 | 286 | 331 | |
| 11 | 160 | 208 | 261 | 313 | 352 | 26 | 160 | 207 | 257 | 303 | 345 | |
| 12 | 143 | 188 | 220 | 273 | 314 | 27 | 169 | 216 | 261 | 295 | 333 | |
| 13 | 154 | 200 | 244 | 289 | 325 | 28 | 157 | 205 | 248 | 289 | 316 | |
| 14 | 171 | 221 | 270 | 326 | 358 | 29 | 137 | 180 | 219 | 258 | 291 | |
| 15 | 163 | 216 | 242 | 281 | 312 | 30 | 153 | 200 | 244 | 286 | 324 |
y <- read.table('https://raw.github.com/wiki/stan-dev/rstan/rats.txt', header = TRUE)
x <- c(8, 15, 22, 29, 36)
xbar <- mean(x)
N <- nrow(y)
T <- ncol(y)
rats_fit <- stan(file = 'https://raw.githubusercontent.com/stan-dev/example-models/master/bugs_examples/vol1/rats/rats.stan')開発チームがBUGS example(WinBUGSに付属している例)の大部分やその他のモデルをStanで作成しており、以下を実行することで試すことができます。
model <- stan_demo()このあとはモデルの例をポップアップで出てきたリストから選んでいきます。はじめてstan_demo()を呼ぶときは、これらの例をダウンロードするかどうかを聞いてきます。option 1を選んでrstanがインストールされたディレクトリに保存するようにしましょう。そうすると、またいつか実行する時に再ダウンロードしなくてもすみます。含まれるmodelオブジェクトはstanfitクラスのインスタンスなので、それらに対して、 print・plot・pairs・extractを呼び出すことができます。
RStanに関するさらなる詳細はrstanパッケージのvignetteに含まれるドキュメントで見ることができます。例えば、help(stan)やhelp("stanfit-class")を使うことでstan関数やS4クラスであるstanfitのヘルプを見ることができます。
StanのサンプラーやオプティマイザーやStanの文法に関する詳細を知りたい時にはStan's modeling language manualを見てください。
さらに、Stanの使い方を議論したい場合にはStan Discourse Forumに例を投稿したり、(R)Stanについて質問してもよいでしょう。助けが必要な時は、以下に関する十分な情報もあわせて記述することが重要です。
- Stanのモデルコード
- データ
- 実行するのに必要なRコード
-
stan関数を呼ぶときにverbose=TRUEかつcores=1を指定して出力されたエラーメッセージのdump - C++コンパイラのバージョン。もし
gccを使っているならばg++ -vで得ることができます。 - R上で
sessionInfo関数で得ることができるRに関する情報
- Gelman, A., Carlin, J. B., Stern, H. S., and Rubin, D. B. (2003). Bayesian Data Analysis, CRC Press, London, 2nd Edition.
- Stan Development Team. Stan Modeling Language User's Guide and Reference Manual.
- Gelfand, A. E., Hills S. E., Racine-Poon, A., and Smith A. F. M. (1990). "Illustration of Bayesian Inference in Normal Data Models Using Gibbs Sampling", Journal of the American Statistical Association, 85, 972-985.
- Stan
- R
- BUGS
- OpenBUGS
- JAGS
- Rcpp